ImageJ: 細胞核のカウント
Materials
Protocol
- ImageJ を起動する。
- 「File」→「Open...」で解析したい画像を開く。

- 「Image」→「Type」→「16-bit」で画像を変換する。

- 「Image」→「Adjust」→「Threshold...」を選択する。
- 「Threshold」ウィンドウが表示される。
左のプルダウンメニューから「Default」を選択する。
右のプルダウンメニューから「B&W」を選択する。
「Dark background」をチェックする。
「Stack histogram」はチェックしない。
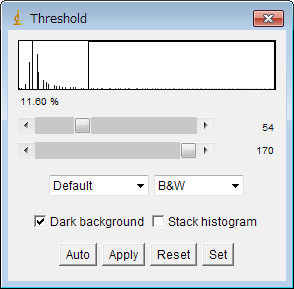
- 「Auto」ボタンを押すと、細胞核が黒く塗り潰される。
ウィンドウのヒストグラムを見ながら、スクロールバーで閾値を調節する。
「Apply」ボタンを押す。

- 「Process」→「Binary」→「Watershed」で、連続した細胞核を分割する。

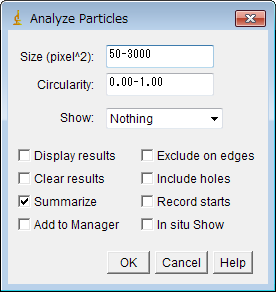
- 「Analyze」→「Analyze Particles...」を選択する。
- 「Analyze Particles」ウィンドウが表示される。
「Size (pixel^2):」にカウントしたいピクセルの面積 (下限-上限) を入力する。
EVOS の ×10 対物レンズで撮影した画像の場合、マウス筋芽細胞で 20-2000 程度、ニワトリ筋芽細胞で 50-3000 程度。
「Summarize」をチェックする。
「OK」ボタンを押す。
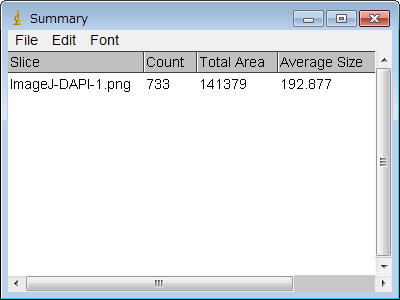
- 「Summary」ウィンドウに結果 (「Count」の数) が表示される。
References








